※온라인 안심존 : 보안이 보장된 온라인 네트워크를 통해 집, 연구실, 사무실 등 어디서나 접속하여 데이터에 접근하고 분석
-
데이터 변경이력
데이터 변경이력 버전 일자 변경내용 비고 1.0 2022-07-13 데이터 최초 개방 데이터 히스토리
데이터 히스토리 일자 변경내용 비고 2024-06-17 산출물 추가 공개 AI모델 2022-07-13 콘텐츠 최초 등록 소개
뇌전이암의 조영증강 뇌 MRI를 중심으로 뇌 CT 및 MRI를 이용하여 뇌전이암의 정확한 위치와 크기를 인식하기 위한 AI 시스템을 학습 시킬 수 있는 데이터셋 구축이 목표임
구축목적
◦ AI 기반 뇌전이암 진단을 위한 다중모델 뇌 영상 데이터 구축 ◦ 뇌전이암 환자의 방사선 치료 설계 보조 인공지능 개발 활용
-
메타데이터 구조표 데이터 영역 헬스케어 데이터 유형 이미지 데이터 형식 dcm 데이터 출처 대학병원 라벨링 유형 세그멘테이션 라벨링 형식 JSON, CSV 데이터 활용 서비스 의료 보조 서비스 데이터 구축년도/
데이터 구축량2021년/1000case, 633,3510slice, 288GB -
데이터 구축 규모
데이터 구축 규모 구분 합계 1차경로 치료전MRI 치료후MRI 치료전CT T1E T2 T1E T2 CT CTE 업로드 이미지(slice) 633,510 159,139 32,084 71,363 17,287 209,900 143,737 *비영상 데이터 1,000 1,000 case 1,000 1,000 *비영상 데이터의 종류 ( 나이, 성별, 종양 개수, 종양 용적, 원발암 ,방사선치료 기법, 방사선치료 용량, 방사선 치료 횟수 ,반응평가 )데이터 분포
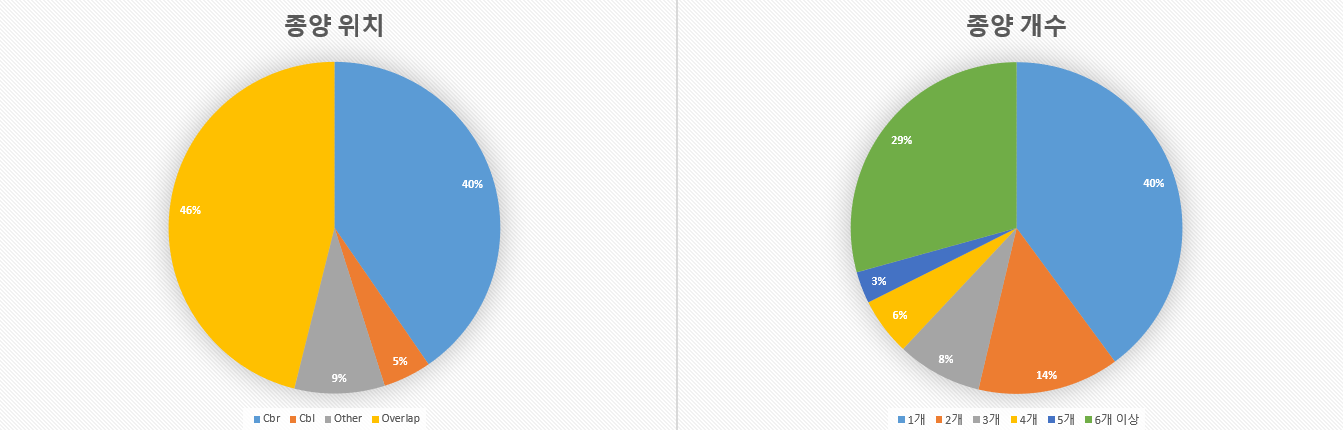
종양위치 케이스 Cbr 404 40.40% Cbl 47 4.70% Other 88 8.80% Overlap 461 46.10% 종양개수 케이스 1개 399 39.90% 2개 138 13.80% 3개 83 8.30% 4개 56 5.60% 5개 31 3.10% 6개 이상 293 29.30% 데이터 분포 그래프
-
-
AI 모델 상세 설명서 다운로드
AI 모델 상세 설명서 다운로드 AI 모델 다운로드학습 알고리즘 RetinaNet 및 U-net 기반
RetinaNet과 U-Net을 함께 사용 또는 U-Net만 사용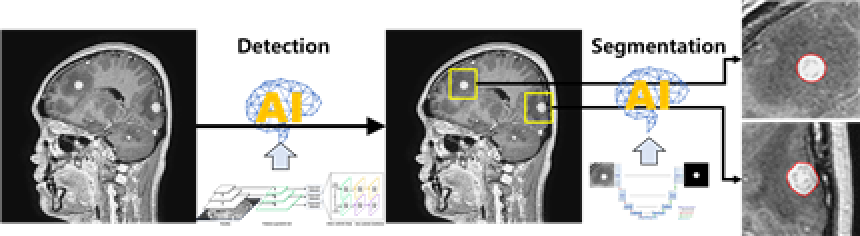
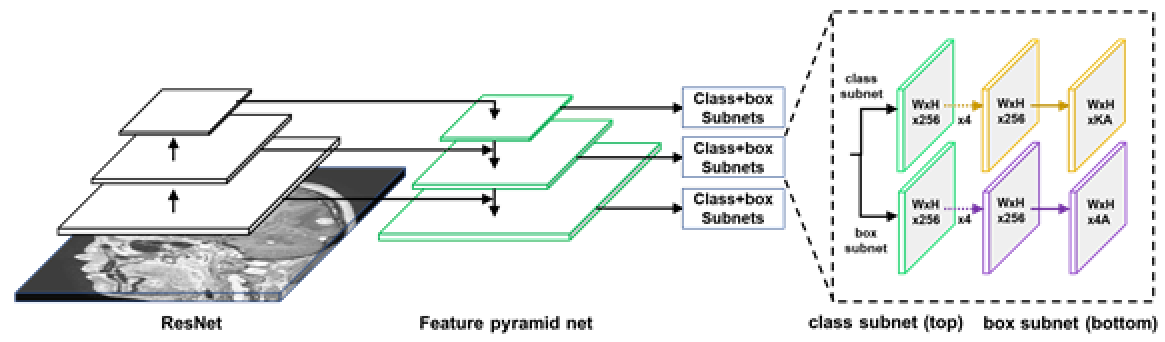
RetinaNet network 개념도
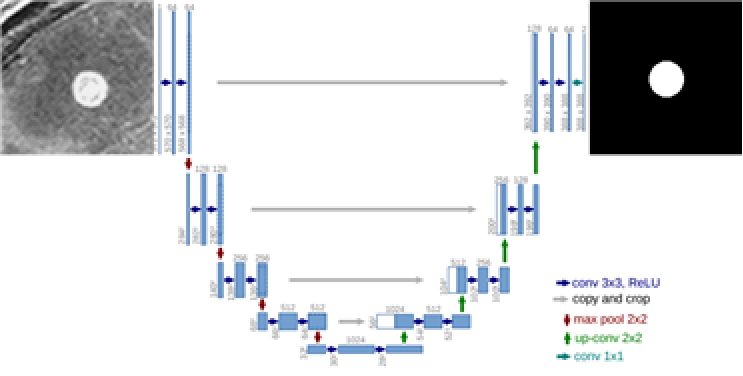
U-net network 개념도
학습 조건 epoch: 500
batch: 128
optimizer: Adam
loss: categorical cross-entropy, focal-loss
iteration: 4,434
*모델 개발 완료 후 파라메터 수치가 변경 될 수 있음.파일 형식 • 학습 데이터셋: DICOM, JSON • 평가 데이터셋: DICOM, JSON 전체구축 데이터 대비 모델에 적용되는 비율 AI모델 사용 이미지 비율(수량)
- 클래스 1: 100% (159,136장)
※ 유효성 검증은 구축된 데이터 전체를 적용하며, 변경이 필요한 경우 TTA 담당자와 협의한다. -
데이터 성능 점수
측정값 (%)기준값 (%)데이터 성능 지표
데이터 성능 지표 번호 측정항목 AI TASK 학습모델 지표명 기준값 점수 측정값 점수 1 종양 분할성능 Segmentation RetinaNet 및 U-net 기반 DSC 0.85 단위없음 0.904899999999999 단위없음 2 종양 탐지 성능 Object Detection RetinaNet 및 U-net 기반 Sensitivity 85 % 86.16 %
※ 데이터 성능 지표가 여러 개일 경우 각 항목을 클릭하면 해당 지표의 값이 그래프에 표기됩니다.
※ AI모델 평가 지표에 따라 측정값의 범위, 판단 기준이 달라질 수 있습니다. (ex. 오류율의 경우, 낮을수록 좋은 성능을 내는 것으로 평가됩니다)
-
설명서 및 활용가이드 다운로드
데이터 설명서 다운로드 구축활용가이드 다운로드구분 내용 구축목적 병변 검출(Detection), 영역 분할(Segmentation) 라벨링 방법 폴리곤 데이터 종류/형식 • 원천 데이터: 영상 DICOM (확장자: dcm)
• 라벨 데이터: JSON, CSV클래스 수량 1종 데이터 실제 예시 • 원천 데이터
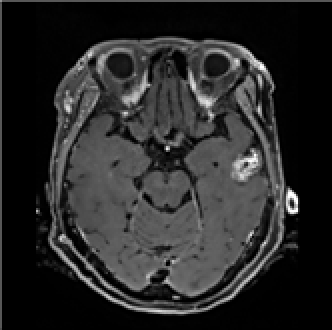
Enhaced T1 MRI• 라벨 데이터
{ "info": {
"id": "GMC-0537",
"date_created": "2015-04-04 },
"patient": {
"age": 63,
"sex": "M",
"primary": "Lung"},
"images": {
"modality": "MR-PRE T1e",
"width": 256,
"height": 256,
"cnt": 164,
"file_name": "GMC-0537",
"slicethickness": 1.0,
"pixelspacing": 0.9960929751396179},
"labels": {
"Total_count": 1,
"Label[1]": {
"Number": 1,
"ROI_type": "free hand",
"volume": 1319.627616112919,
"B_box_z_minmax": [107, 119],
"B_box_y_minmax": [115, 127],
"B_box_x_minmax": [107, 119],
"coordinates_(z,y,x)": [
[107,117,82],
... ...라벨링 구성요소
라벨링 구성요소 구분 항목명 타입 필수여부 설명 범위 비고 1 info object 데이터셋정보 1-1 info.id string M 데이터 식별자 1-2 info.date_created string O 촬영일자 YYYY-MM-DD 2 patient object 환자정보 2-1 patient.age number M 나이 [1~100] 2-2 patient.sex string M 성별 F, M F: 여자, M: 남자 2-3 patient.primary string M 원발암 [lung,colorectum,breast,ns,pns,biliary system, , ...]
*전체 범위는 합의서 p.??에 상세 기제3 images object M 이미지정보 3-1 images[].modality string M 이미지모달리티 MR-PRE T1e,
MR-PRE T2 MR-Post T1e,
MR-Post T2,
CT, Cte3-2 images[].width number M 이미지 너비 [1~2048] 3-3 images[].height number M 이미지 높이 [0~2048] 3-4 images[].cnt number M 이미지 개수 [1~500] 3-5 images[].file_name string M 이미지 파일명 3-6 images[].slicethickness number M 슬라이스 두께 [0.0001~10] mm 3-7 images[].pixelspacing number M 픽셀 간격 [0.0001~1.5] mm 4 labels object O 라벨 정보 4-1 labels.Total_count number M 전체 라벨 개수 [1~1000] 4-2 labels.Label array 4-2-1 labels.Label.Number number M 라벨 번호 [1~1000] 라벨번호 4-2-2 labels.Label.ROI_type string M ROI의 기하학적 속성 free hand ROI의 기하학적 속성 4-2-3 labels.Label.volume number M 라벨체적 - 개별 json파일에서 제공 4-2-4 labels.Label.B_box_z_minmax array M 라벨에 대한 바운딩박스 [0~500] z 4-2-5 labels.Label.B_box_y_minmax array M 라벨에 대한 바운딩박스 [0~500] y 4-2-6 labels.Label.B_box_x_minmax array M 라벨에 대한 바운딩박스 [0~500] x 4-2-7 labels.Label.coordinates_(z,y,x) array M 라벨 위치좌표 [0~500] 각각
x,y,z임상정보 데이터(CSV)
구분 항목명 타입 필수
여부설명 범위 비고 CRF No string Y 데이터 번호 GMC-0001~
GMC-0914,
INH-0001~
INH-0311DateCT1 string Y CT촬영날짜 Age number Y 나이 [1-120] Sex string Y 성별 F: 여자,
M: 남자F:여자, M:남자 Primary string Y 원발암 [lung,colorectu
m,breast,ns,pns,
biliary system,
...]
* primary의 범위
(유효값) 참조RT_tech string Y 방사선치료기법 "2D", "3D CRT", "CK", "IMRT", "RapidArc", "SRS" MR_Date string 치료 후 MR 촬영날짜 RT_eff string 반응평가 "CR","PD","PR",
"SD","X"X: 반응평가 없음 T_loc number Y 종양 위치 [1~4] 1.Cbr, 2.Cbl, 3.Other, 4.Overlap T_num number Y 종양 개수 [1~6] 6: >5개 T_vol number Y 종양 용적 [0~500] cubic cm RT_crs number Y 치료차수 [1~5] 회 RT_dose number Y 조사량 [1~10000] Gy RT_fx number Y 치료횟수 [1~30] 회 *primary의 범위 (유효값)
"Astrocytoma", "Biliary System", "Bladder", "Bone Mets", "Brain", "Brain Mets", "Breast", "Cervix", "Colorectum", "Corpus", "Esophagus", "Germ Cell", "Hypopharynx", "Larynx", "Leukemia", "Lip & Oral", "Liver", "Lung", "Melanoma", "Nasopharynx", "NC & PNS", "NHL", "NS", "OS & STS", "Other CNS", "Other GO", "Other Sarcoma", "Other Skin", "Other UR", "Pancreas", "Prostate", "Stomach", "Rectum", "Thyroid", "Unk Prim H&N", "Unk Primary", "Biliary, GB", "breast", "Colon", "Colon, Rectum", "Anus", "Kidney, adrenal", "Kidney, RCC", "others", "Ovary, adnexa", "Renal pelvis, RCC", "Skin", "Small intestine", "Testis", "Uterus", "Colon, Rectum, Anus"
-
데이터셋 구축 담당자
수행기관(주관) : 가천대학교 산학협력단
수행기관(주관) 책임자명 전화번호 대표이메일 담당업무 이 언 032-460-3304 [email protected] AI모델, 데이터 라벨링 김광기 070-7120-7912 [email protected] 수행기관(참여)
수행기관(참여) 기관명 담당업무 ㈜브라이센코리아 데이터 라벨링
-
인터넷과 물리적으로 분리된 온라인·오프라인 공간으로 의료 데이터를 포함하여 보안 조치가 요구되는 데이터를 다운로드 없이 접근하고 분석 가능
* 온라인 안심존 : 보안이 보장된 온라인 네트워크를 통해 집, 연구실, 사무실 등 어디서나 접속하여 데이터에 접근하고 분석
* 오프라인 안심존 : 추가적인 보안이 필요한 데이터를 대상으로 지정된 물리적 공간에서만 접속하여 데이터에 접근하고 분석 -
- AI 허브 접속
신청자 - 안심존
사용신청신청자신청서류 제출* - 심사구축기관
- 승인구축기관
- 데이터 분석 활용신청자
- 분석모델반출신청자
- AI 허브 접속
-
1. 기관생명윤리위원회(IRB) 심의 결과 통지서 [IRB 알아보기] [공용IRB 심의신청 가이드라인]
2. 기관생명윤리위원회(IRB) 승인된 연구계획서
3. 신청자 소속 증빙 서류 (재직증명서, 재학증명서, 근로계약서 등 택1)
4. 안심존 이용 신청서 [다운로드]
5. 보안서약서 [다운로드]
※ 상기 신청서 및 첨부 서류를 완비한 후 신청을 진행하셔야 정상적으로 절차가 이루어집니다. -
신청 및 이용관련 문의는 [email protected] 또는 02-525-7708, 7709로 문의
데이터셋 다운로드 승인이 완료 된 후 API 다운로드 서비스를 이용하실 수 있습니다.
API 다운로드 파일은 분할 압축되어 다운로드 됩니다. 분할 압축 해제를 위해서는 분할 압축 파일들의 병합이 필요하며 리눅스 명령어 사용이 필요합니다.
리눅스 OS 계열에서 다운로드 받으시길 권장하며 윈도우에서 파일 다운로드 시 wsl(리눅스용 윈도우 하위 시스템) 설치가 필요합니다.
※ 파일 병합 리눅스 명령어
find "폴더경로" -name "파일명.zip.part*" -print0 | sort -zt'.' -k2V | xargs -0 cat > "파일명.zip"
- 해당 명령어 실행 시, 실행 경로 위치에 병합 압축 파일이 생성됩니다.
- 병합된 파일 용량이 0일 경우, 제대로 병합이 되지 않은 상태이니 "폴더경로"가 제대로 입력되었는지 확인 부탁드립니다.
※ 데이터셋 소개 페이지에서 다운로드 버튼 클릭하여 승인이 필요합니다.